X染色体不活化の安定性は染色体の形が鍵だった -半世紀以上の時を経て、不活性X染色体の複製の謎が明らかに-

理化学研究所(理研)生命機能科学研究センター発生エピジェネティクス研究チームのラウィン・プーンパーム研究員、平谷伊智朗チームリーダー、近畿大学農学部生物機能科学科動物分子遺伝学研究室の佐渡敬教授、大阪大学大学院理学研究科生物科学専攻染色体構造機能学研究室の小布施力史教授らの共同研究グループは、哺乳類の雌の「不活性X染色体[1]」の特徴的なDNA複製[2]制御の解析から、その3次元構造に関する新しい特徴を見いだしました。
本研究成果は、1960年に見いだされた不活性X染色体のS期[3]後期複製の意義に初めて言及し、染色体[4]構造と遺伝子発現[5]制御の関係を明らかにするもので、高度に凝縮したヘテロクロマチン[6]と呼ばれる染色体構造が遺伝子発現を安定的に抑制する仕組みの理解につながると期待できます。
雌の細胞が持つ2本のX染色体のうち1本は胚発生の初期に不活性化され、不活性X染色体となって遺伝子発現が抑制されます。不活性X染色体の複製様式は他の染色体とは異なり、細胞周期[3]のS期後期に複製されます。
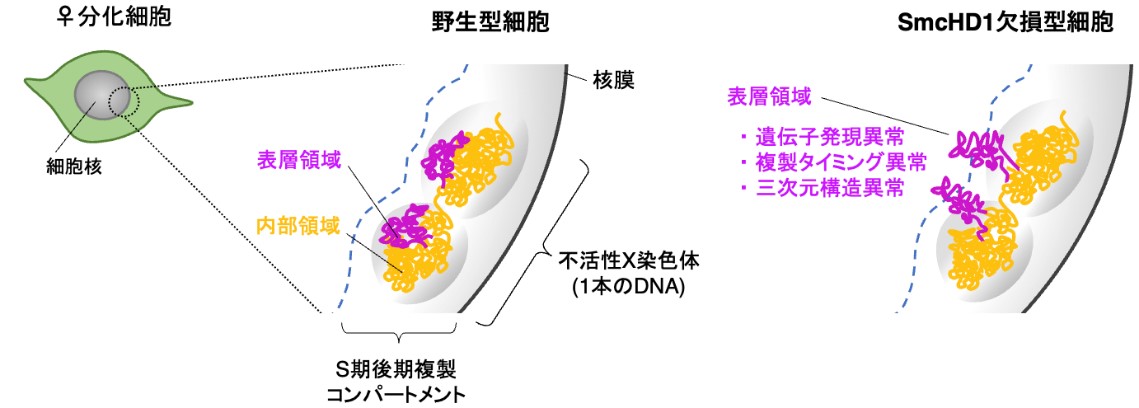
今回、共同研究グループは、マウスES細胞[7]の分化に伴う不活性X染色体の複製タイミング[2]の変化と3次元構造の変化、不活性X染色体結合タンパク質SmcHD1の役割を調べました。その結果、(1)不活性X染色体全域がS期後期に複製されるのは高度に凝縮した染色体構造をよく反映していること、(2)不活性X染色体は一見均一に凝縮しているように見えるが、染色体テリトリー[4]内部に層構造が存在していること、(3)SmcHD1が不活性X染色体の表層領域の不活性化状態、適切な複製タイミング、および3次元構造の維持に必要であることなどを明らかにしました。
本研究は、科学雑誌『Nature Structural and Molecular Biology』オンライン版(8月10日付:日本時間8月11日)に掲載されました。
【背景】
哺乳類の性染色体の構成は、雄はXY、雌はXXであり、雌は2本のX染色体を持っています。Y染色体には、精子形成や性決定に関わる遺伝子が50個ほどあるだけなので、哺乳類の生存にはY染色体の有無はあまり影響しません。しかし、X染色体には生存に不可欠なものも含めて1,000個ほどの遺伝子があります。そのため、雌雄間でX染色体の遺伝子量の差を補正するメカニズムを進化させたと考えられています。それが「X染色体不活性化」です。すなわち、胚発生の初期に雌は2本あるX染色体のうちの1本を不活性化し、機能するX染色体を雄と同じ1本になるようにします。不活性化されたX染色体を「不活性X染色体」(以下、不活性X)といいます。
不活性Xは、条件的ヘテロクロマチン[6]の代表例であり、クロマチン高次構造と遺伝子発現制御の関係を理解する上での格好の研究モデルです。これまで、X染色体不活性化に関わるRNAやタンパク質がたくさん見つかっていますが、不活性Xのヘテロクロマチン構造、さらにはその3次元構造がどのように構築されるのかはよく分かっていませんでした。
哺乳類の染色体は、細胞周期の間期[3]の核内空間でそれぞれ独立した空間を占め、その内部では、数珠つなぎになったトポロジカルドメイン(TAD)[8]と呼ばれる約1Mb(メガベース、100万塩基対)の染色体ドメインが、各々の性質に応じてAコンパートメント[9]もしくはBコンパートメント[9]を形成し、空間的にまとまって存在すると考えられています。しかし、不活性Xの3次元構造をHi-C[10]で解析しても、TADとA/Bコンパートメントらしきものは存在しません。
また不活性Xは、特殊な複製様式をとることも知られています。他の染色体では、細胞周期のS期初期に複製される領域とS期後期に複製される領域が混在しているのに対し、不活性Xは染色体全体がS期後期に複製される様子が1960年に明らかにされています。そのため、S期後期の複製は不活性Xの指標として長らく重宝されてきましたが、実はその生物学的意味はよく分かっていません。最近の研究から、複製タイミングがA/Bコンパートメント構造をかなりよく反映していることが明らかになってきました。複製タイミングの違いは、直接的にはゲノム領域ごとの複製開始タンパク質の結合しやすさの違いを反映していると考えられていますが、不活性Xでもこの相関が成り立つかは分かっていません。
そこで共同研究グループは、不活性Xの複製タイミングの制御を詳しく調べることで、不活性Xの3次元構造、特にコンパートメント構造について新たな知見が得られると考えて研究に着手しました。
【研究手法と成果】
不活性Xの形成過程の観察には、マウスES細胞の分化系が有効です。雌由来のES細胞は、未分化状態では不活性Xは見られませんが、分化が誘導されるとX染色体のどちらかがランダムに不活性化されます。共同研究グループは、2本のX染色体が一塩基多型(SNP)[11]で識別でき、さらに2本のうち1本が選択的に不活化されるように工夫された特別なES細胞を実験に用いました。このES細胞を、未分化段階、分化後7~9日目の神経外胚葉、分化後3週間たった神経幹細胞の三つの分化段階に分けて、複製タイミングを解析しました。
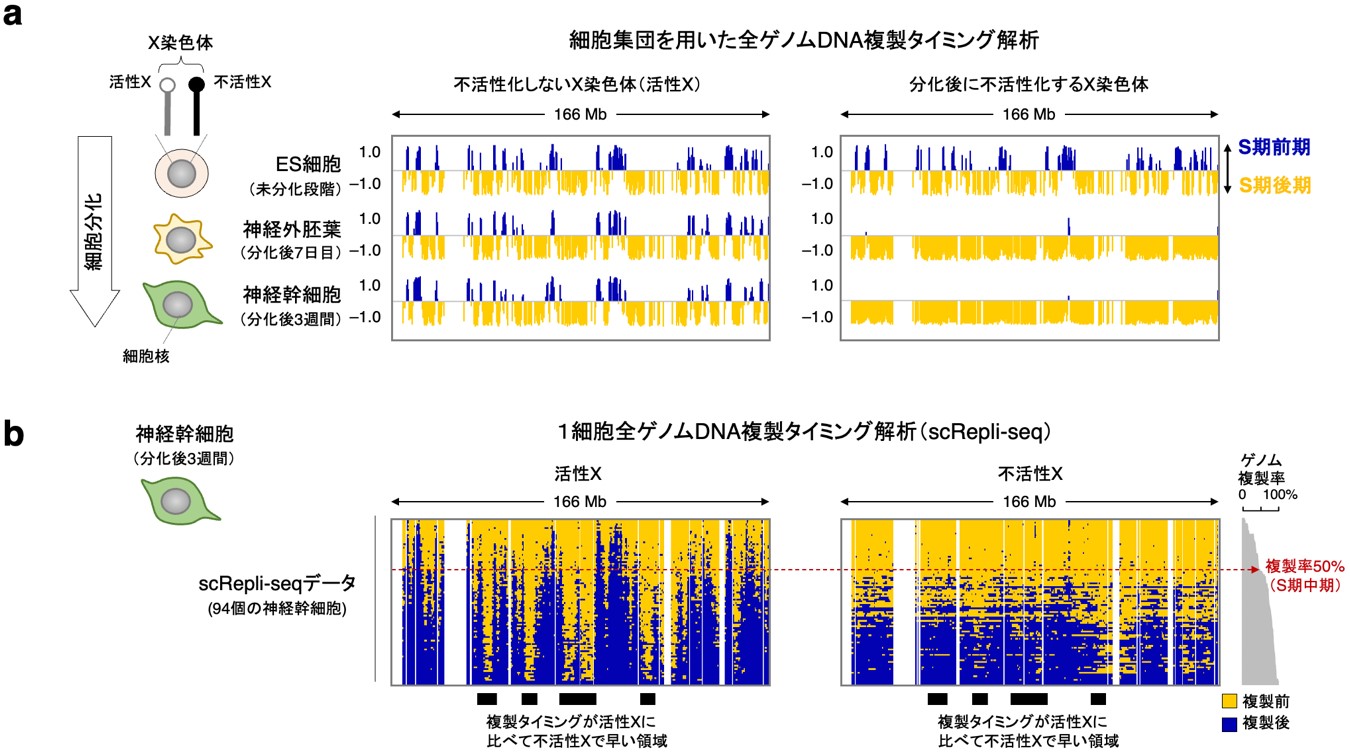
まず、1万個ほどの細胞集団の平均的な複製タイミングを全ゲノムDNA複製解析法(Repli-seq[12])で調べたところ、分化後7日目に不活性Xの染色体全域がS期後期に複製されることが分かりました(図1a)。次に、独自に開発した1細胞全ゲノムDNA複製解析法注1)(scRepli-seq[12])を用いて、一つ一つの神経幹細胞ごとに複製タイミングを解析しました。すると、不活性X全域がS期後期に複製されるのは細胞集団の解析と同じ結果でした。しかし、よく見ると不活性X上に、不活性化しないX染色体(以下、活性X)に比べて複製タイミングが早い領域も認められ、染色体全域で複製タイミングが均一化(不活性Xの領域間で複製される時期にほとんど差が見られない)していました(図1b)。つまり、ES細胞の分化に伴って、不活性Xが複製を開始するタイミングは細胞ごとにばらつくものの、いったん複製が始まるとそのプロセスは数時間で一気に完了することが分かりました。

a)マウスES細胞と、ES細胞を分化させて生じた神経外胚葉および神経幹細胞を用いた、細胞集団全ゲノムDNA複製タイミング解析(Repli-seq)データ。不活性Xは、分化後7日目には染色体全域がS期後期複製を示す(右)一方、不活性化しない活性Xは分化の過程を通して複製タイミングをほぼ変化させない(左)。
b)マウス神経幹細胞を用いた1細胞全ゲノムDNA複製タイミング解析(scRepli-seq)データ。S期全域から94個の細胞を回収し、ゲノムDNA複製率(ほとんど未複製なものから、複製完了に近いものまで)に従って上から下に並べたもの。赤の点線は、常染色体の複製率50%で定義したS期中期の細胞を示す。不活性Xの大半は、S期中期を過ぎてから複製を開始する(S期中期以前は、染色体のほぼ全領域が複製前の黄色の状態)。これに対し、活性XではS期初期から複製が開始しており、複製時期が早い領域、遅い領域が明確に分かれている。不活性Xはこのような明確な複製タイミングパターンを示さず、活性Xに比べて複製タイミングが早くなる領域もあちこちに存在する(黒線)。
不活性Xの複製タイミングや核内コンパートメント[9]制御に関わる因子の一つに、不活性X結合タンパク質SmcHD1が知られています。そこで、不活性Xの複製タイミングがどのように制御されているのかの手掛かりを得るために、SmcHD1欠損型ES細胞を作製し、複製タイミングにどのような影響が現れるか調べました。
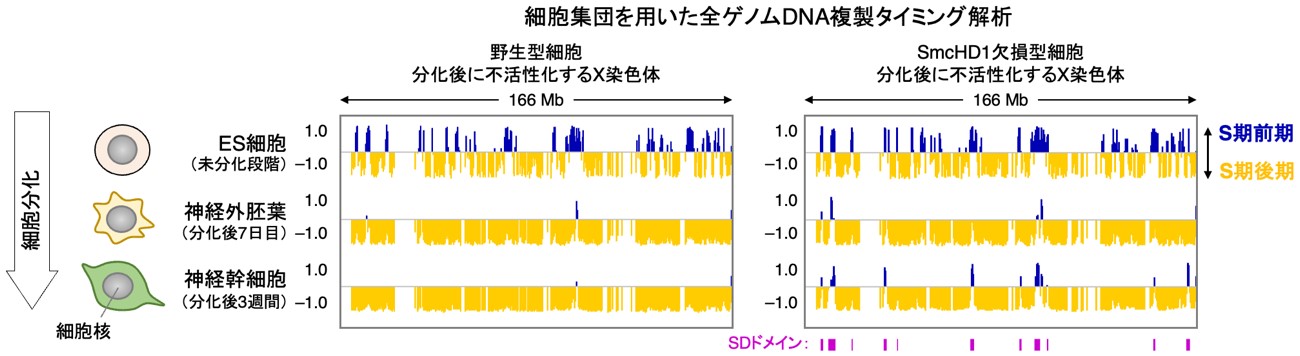
まず、細胞集団を用いた複製タイミングを解析すると、SmcHD1欠損型細胞では、分化が進む(分化後7~9日目)と不活性Xがいったんは染色体全域でS期後期に複製されるようになるものの、神経幹細胞に分化すると一部の領域の複製タイミングが早くなり、不活性XのS期後期複製が維持できなくなることが分かりました(図2)。この現象はSmcHD1欠損型細胞集団中の一部で見られるものではなく、大多数の細胞で見られました。

細胞集団全ゲノムDNA複製タイミング解析で得られたB6由来X染色体(不活性X)データ。SmcHD1欠損型神経幹細胞は、不活性XのS期後期複製を維持できない。そのような領域をSDドメイン(SD:SmcHD1-Dependent[SmcHD1依存的])と名付けた。
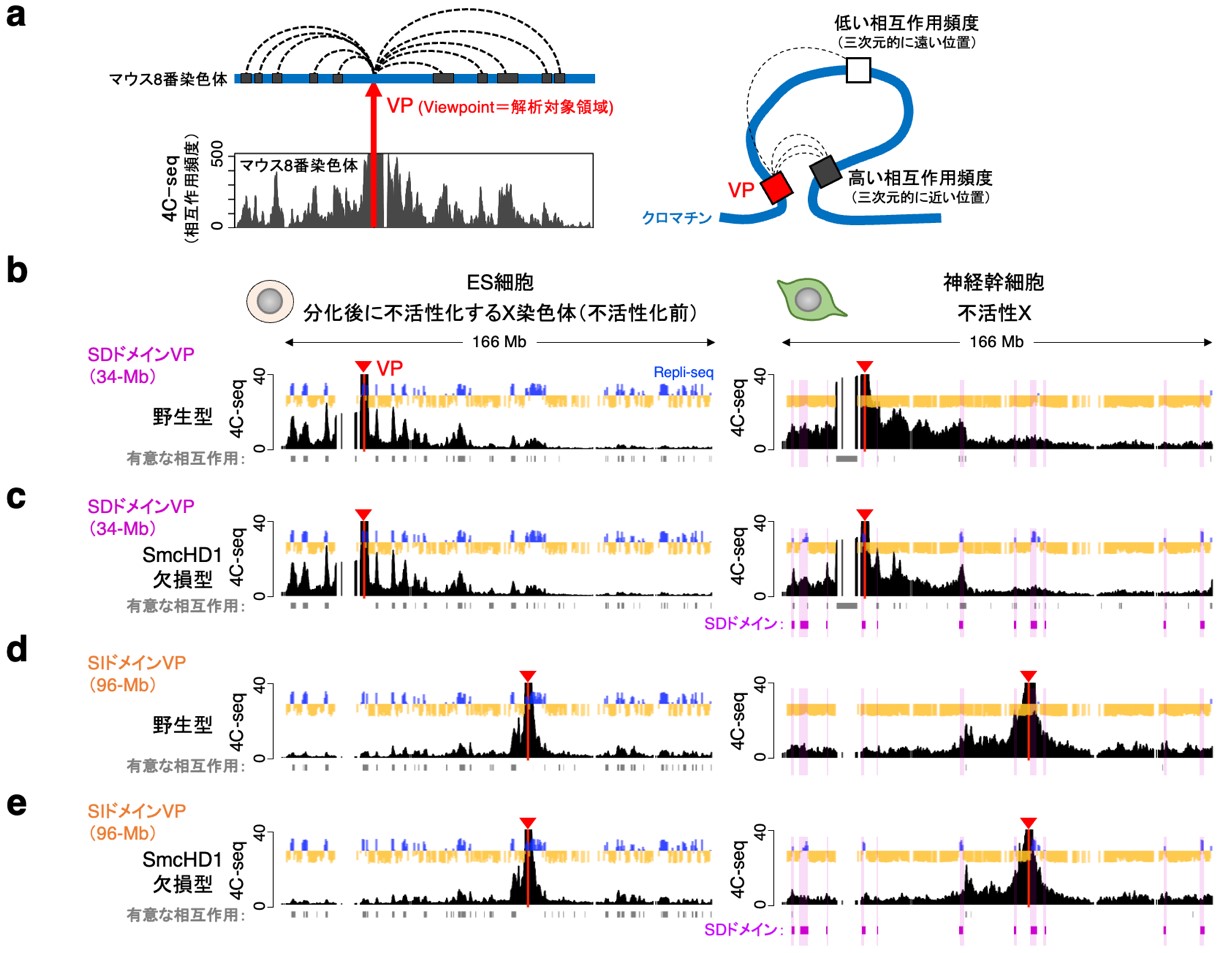
SmcHD1は不活性X全域に結合するとされています。では、なぜSmcHD1欠損型細胞で複製タイミングが早くなる変化はX染色体全域で起きるのではなく、一部の領域に生じるのでしょうか。常染色体上では、複製タイミングと染色体の高次構造(A/Bコンパートメント構造)が強く相関しており、不活性Xでも同様の相関が成り立つ可能性があると考えられます。そこで、SmcHD1欠損型細胞でS期後期複製を維持できなかった不活性X領域をSDドメイン(SD:SmcHD1-Dependent[SmcHD1依存的])、維持できた領域をSIドメイン(SI:SmcHD1-Independent[SmcHD1非依存的])と定義し、両者の高次構造の違いを4C-seq[10]と呼ばれる方法で調べました(図3a)。
その結果、野生型ES細胞では、SDドメインは他のS期前期複製ドメインと強い相互作用を示し、S期後期複製ドメインとはほぼ相互作用しませんでした(図3b左)。しかし、分化が進んで神経幹細胞になると、SDドメインは近隣の染色体領域と満遍なく相互作用するようになりました(図3b右)。この結果は、分化した細胞では、染色体全域で複製タイミングがS期後期に均一化することと一致します。また、SIドメインも、SDドメインとほぼ同じ相互作用パターンを示しました(図3d、ES細胞では他のS期前期複製ドメインと強く相互作用し、分化後の神経幹細胞では近隣の染色体領域と満遍なく相互作用)。
一方、SmcHD1欠損型ES細胞では、SD、SIドメインともに、その相互作用パターンは野生型ES細胞と同じで、S期前期複製ドメインと強い相互作用を示し、S期後期複製ドメインとはほぼ相互作用しませんでした(図3c左、3e左)。しかし、分化が進んだSmcHD1欠損型神経幹細胞では、SDドメイン同士が相互作用している様子が捉えられました(図3c右)。これはSIドメインには見られない特徴でした(図3e右)。

a)4C-seqはVP(Viewpointの略。解析対象領域)と、他のゲノム領域との相互作用頻度を解析するためのHi-Cに類似の全ゲノム解析手法。黒のピークが高い領域は、VPとの強い相互作用を表す(図はマウス8番染色体の解析例で、Dileep et al., Genome Res, 2015を一部改変)。
b)野生型のES細胞(左)と神経幹細胞(右)の4C-seqデータ。4C-seqデータの背景に描かれているのは、図1で示した細胞集団全ゲノムDNA複製タイミング解析データ(Repli-seq)で、青色のピークがS期前期に、黄色がS期後期に複製される領域を示す。VPは34-Mb領域に存在するSDドメイン。このVPは、野生型ES細胞ではS期前期に、分化後の神経幹細胞ではS期後期に複製される。このVPは、野生型ES細胞(左)では他のS期前期複製ドメインと強く相互作用するが、分化後の神経幹細胞(右)では近隣の染色体領域と満遍なく相互作用している。
c)SmcHD1欠損型のES細胞(左)と神経幹細胞(右)の4C-seqデータ。VPは34-Mb領域に存在するSDドメイン。このVPは、SmcHD1欠損型ES細胞でもSmcHD1欠損型神経幹細胞でも、S期前期に複製される。SmcHD1欠損型神経幹細胞(右)の不活性XのSDドメイン(VP以外のマゼンタの領域)が、野生型神経幹細胞(b右)に比べて明らかにVPとの相互作用頻度を上昇させており、有意な相互作用を示す。
d)野生型のES細胞(左)と神経幹細胞(右)の4C-seqデータ。VPは96-Mb領域に存在するSIドメイン。このVPは、野生型ES細胞ではS期前期に、分化後の神経幹細胞ではS期後期に複製される。このVPは野生型ES細胞(左)では他のS期前期複製ドメインと強く相互作用するが、分化後の神経幹細胞(右)では近隣の染色体領域と満遍なく相互作用している。
e)SmcHD1欠損型のES細胞(左)と神経幹細胞(右)の4C-seqデータ。VPは96-Mb領域に存在するSIドメイン。このVPは、SmcHD1欠損型ES細胞ではS期前期に、分化後のSmcHD1欠損型神経幹細胞ではS期後期に複製されるが、dの野生型データとほとんど変わらない4C-seqの結果を示した。
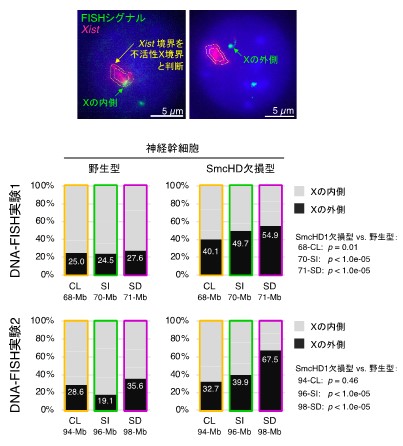
SDドメインとSIドメインの染色体内の相互作用の違いをさらに詳しく調べるため、各ドメインの核内での配置を蛍光顕微鏡で観察しました。間期の核内でX染色体は、核膜に近い独立した空間を占めていますが、SmcHD1欠損型神経幹細胞では、SDドメインが頻繁にX染色体テリトリーの外に飛び出していました(図4)。SIドメインも同じ傾向を示しましたが、飛び出す頻度はSDドメインより低いことが分かりました(図4下)。さらに、これら二つのドメインの比較として、常にS期後期複製するドメインをCLドメイン(CL:Constitutively Late[恒常的に遅い])と定義し観察したところ、SmcHD1欠損型細胞の不活性Xの染色体テリトリーに、表層側からSD、SI、CLドメインと並ぶ層構造が存在していることが示唆されました。

SD、SI、CLドメインの蛍光顕微鏡像による比較解析(DNA-FISH解析)。それぞれのドメインから二つの異なる領域を解析対象に選び、各ドメインが不活性X染色体(不活性Xを覆う非コードRNAであるXistシグナルの境界を不活性Xの境界と判断)の内側か外側どちらに局在しているかを蛍光像(FISHシグナル)の位置で判定した(上の写真)。下は、DNA-FISH実験1と2のグラフで、ドメイン名の下の数字はそれぞれの実験で用いた各ドメインの染色体上の位置を表す。SmcHD1欠損型細胞で不活性Xの外に飛び出ている頻度はSDドメインが最も高く、次いでSI、CLドメインの順に高い。
また、ドメイン間の違いを遺伝子の特徴から調べると、SDドメインは、SIドメインに比べて内部にある遺伝子の再活性化が高頻度で起きており、不活性X特有の抑制型エピジェネティック修飾[13]レベルは低下し、活性型修飾レベルは上昇していました。さらにSDドメインには、不活性Xによる抑制を外れて発現できるエスケープ遺伝子[14]が有意に多いことも判明しました。
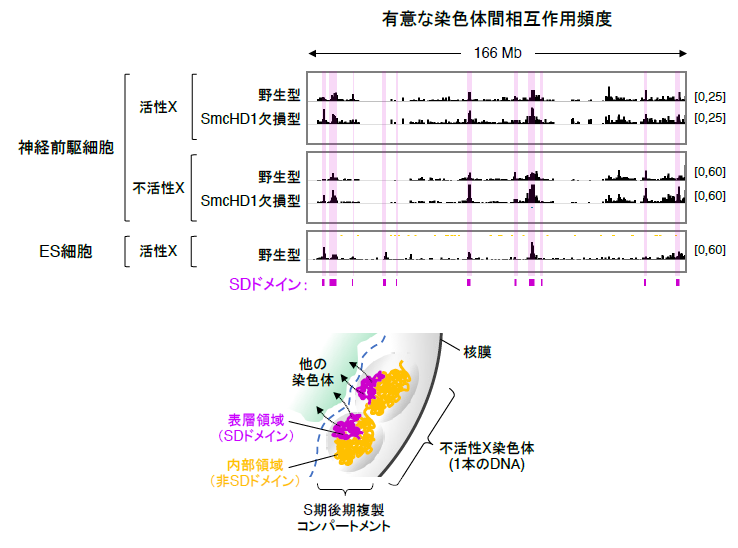
このように、SmcHD1欠損型神経幹細胞では、SDドメインは不活性Xの染色体テリトリーの外に飛び出して相互作用しているらしいことが分かりましたが、この現象は単に遺伝子の再活性化の結果か、それとも原因なのでしょうか。染色体間の相互作用を、SmcHD1欠損型細胞と野生型細胞、さらに未分化ES細胞を含めて改めて調べてみると、不活性XのSDドメインは他の染色体と高頻度で相互作用していることが、不活化の有無にかかわらず観察されました(図5)。すなわち、不活性X上のSDドメインが他の染色体と高頻度で相互作用するのは、細胞の分化状態やX染色体不活性化状態とは関係がなく、SDドメインのDNA配列に固有の性質であることが示唆されました。

野生型かSmcHD1欠損型、活性Xか不活性Xを問わず、ES細胞でも神経前駆細胞でもX染色体上の染色体間相互作用頻度の分布は似通っていた(上)。SDドメインが最も強い染色体間相互作用頻度を示し、これらが不活性X染色体テリトリーの表層領域に対応することが示唆された(下)。Hi-Cデータは、Wang et al., Cell, 2018由来。
以上の結果から、不活性Xの複製タイミングと3次元構造の制御に関して、考えられる最新の知見を次のようにまとめることができました。
(1)不活性Xが形成される際、不活性X全域がS期後期に複製されるが、これは不活性Xが高度に凝縮している状態を反映している。
(2)不活性Xは一見均一に凝縮しているように見えるが、その染色体テリトリー内部にはおそらく層構造が存在する。
(3)SmcHD1は、不活性Xの最表層の不活化状態および正常な複製タイミングと3次元構造の維持に必要である。
(4)不活性Xの最表層は、そもそも遺伝子が再活性化されやすい性質を持っており、不活化を免れるエスケープ遺伝子が濃縮されている。
注1)2019年2月26日プレスリリース「ゲノムDNA複製の真の姿を捉えた」
https://www.riken.jp/press/2019/20190226_1/index.html
【今後の期待】
本研究により、不活性X染色体の特殊な複製様式の意義とその3次元構造に関する理解が一歩進んだことになります。特定のゲノム3次元構造が転写活性の原因か結果かについては長い間議論されてきました。今回の発見は、ゲノム3次元構造がゲノム機能に対して因果的な役割を果たすことを示唆するもので、ゲノム3次元構造の生物学的意義に言及できる貴重な結果であると考えています。また、今回得られたデータは、不活性X染色体内部にこれまで知られていなかった層構造が存在することを示唆しています。共同研究グループは、今後、この仮説の妥当性をさらに検討するとともに、不活性Xの特殊な3次元構造が生体内でどのように構築されていくかという課題にも取り組みたいと考えています。
SmcHD1欠損型細胞のHi-C解析論文はこれまでに複数発表されていますが、不活性X上のSDドメインの挙動に言及した報告はありません。そこで、今回改めて先行研究のHi-Cデータを共同研究グループが再解析したところ、既存のSmcHD1欠損型細胞Hi-CデータにおいてもSDドメイン間の相互作用が見えていたにもかかわらず、単に見落とされていただけであったことが分かりました。つまり、現在公開されているHi-Cデータには、他にも通常のHi-C解析からは見えてこない"宝物"がまだ隠れている可能性があるのではないかと考えています。
【論文情報】
<タイトル>
Replication dynamics identifies the folding principles of the inactive X chromosome
<著者名>
Rawin Poonperm, Saya Ichihara, Hisashi Miura, Akie Tanigawa, Koji Nagao, Chikashi Obuse, Takashi Sado, Ichiro Hiratani
<雑誌>
Nature Structural and Molecular Biology
<DOI>
10.1038/s41594-023-01052-1
【補足説明】
[1]不活性X染色体
X染色体不活性化を受けて遺伝子発現が抑制されたX染色体のこと。哺乳類の雌の細胞では、胚発生の初期に2本のX染色体のうち1本の染色体が不活性化されて、高度に凝縮した3次元構造をとる。これにより、X染色体からの遺伝子の発現量が雄と雌の間で同じになるように調節(遺伝子量補償)されている。
[2]DNA複製、複製タイミング
DNA複製は、ゲノムDNA複製、ゲノム複製と同義。細胞分裂における核分裂の前に、DNAポリメラーゼという酵素によって、ゲノムDNAが過不足なく倍加される過程。なお、ゲノムDNAとは、生物の細胞が持っている全てのDNA配列情報のことを指し、真核生物の場合は核内にある染色体DNAを指すことが多い。複製タイミングとは、ゲノムDNA複製の時間的制御のこと。S期において、各ゲノムDNA領域は、それぞれ特徴的な時期に複製されている。
[3]S期、細胞周期、間期
増殖中の真核細胞が繰り返す時間的なサイクル(期間)のことを細胞周期と呼ぶ。細胞周期の中で、分裂期(M期)は細胞分裂を行う期間、間期は分裂期以外の期間を指す。間期は、さらにG1期、S期、G2期に分けられる。S期は、DNA複製を行う期間である。
[4]染色体、染色体テリトリー
染色体は二倍体の生物の細胞核内に存在し、DNAとタンパク質などから成る構造体で遺伝物質の本体。ヒトの細胞は、1番から22番染色体まで22対の父方由来、母方由来の相同染色体を持ち、これに加えて2本の性染色体を持つ(雄はXY、雌はXX)。細胞周期の間期の核内空間において、各々の染色体が占有する領域を染色体テリトリーと呼ぶ。
[5]遺伝子発現
ゲノムDNA上の遺伝子配列を基にRNAが合成されることを転写、転写されたRNAがタンパク質に変換されることを翻訳と言う。このようにゲノムDNA上の遺伝子配列情報が、転写、翻訳のプロセスを経て細胞における構造や機能に変換される一連の過程のことを遺伝子発現と言う。
[6]ヘテロクロマチン、条件的ヘテロクロマチン
クロマチンとは、真核生物の核内でゲノムDNAが形成する高次構造体のこと。その主成分はDNAとヒストンと呼ばれるタンパク質。ヘテロクロマチンとは、高度に凝縮して遺伝子発現活性が弱い領域のクロマチン構造のこと。ヘテロクロマチンには、大別して、構成的ヘテロクロマチンと条件的ヘテロクロマチンがあるとされており、前者は細胞種を問わず安定的に存在するが、後者は、発生・分化の過程で新たに形成されたり消失したりする。
[7]ES細胞
哺乳類生物の発生初期の胚盤胞期と呼ばれる時期の胚の一部、内部細胞塊と呼ばれる細胞群から作製する幹細胞株のこと。胚性幹細胞とも呼ばれる。ESはEmbryonic Stemの略。
[8]トポロジカルドメイン(TAD)
Hi-Cによって見いだされた染色体上に存在する約1Mb長の染色体ドメインのこと。相互作用頻度の高い一つながりのDNA領域に対応しており、TAD間には相互作用のギャップ(境界)が存在する。TADは、異なる細胞種間でも高度に保存されている普遍的な構造と考えられている。TADはTopologically Associating Domainの略。
[9]A/Bコンパートメント、核内コンパートメント
類似の性質を持つTAD同士は細胞核内で寄り集まって、核内でコンパートメントという相互排他的な(混ざり合わない)領域を核内空間に形成している。核内コンパートメントは性質に応じて2つに大別され、Aコンパートメントはよく転写されていて、複製タイミングがS期前半の領域(ユークロマチン)とよく一致し、Bコンパートメントは転写が抑えられていて、複製タイミングがS期後半の領域(ヘテロクロマチン)とよく一致する。
[10]Hi-C、4C-seq
Hi-C、4C-seqともに3C(Chromosome Conformation Capture)法を発展させた全ゲノム解析手法のこと。Hi-Cは、細胞核内3次元空間におけるあらゆるゲノムDNA配列同士の相対距離を測定して、染色体の3次元的な構造を推定できる画期的な手法。4C-seqは、Hi-C以前に開発された手法であり、特定ゲノム領域対全ゲノム領域の相対距離を測定し、3次元構造を推定する手法。
[11]一塩基多型(SNP)
ある生物種集団のゲノムDNA配列中に1塩基が変異した多様性が見られ、その変異が集団内で1%以上の頻度で見られるとき、これを一塩基多型という。塩基とは核酸の構成要素のことで、DNAの場合はアデニン(A)、チミン(T)、グアニン(G)、シトシン(C)の4種類を指す。実際には、1塩基に限らず、2塩基から十数塩基程度の短い置換、挿入、欠失などの変異も含めて一塩基多型と呼ぶことも多く、本研究でもこの立場をとっている。SNPはSingle Nucleotide Polymorphismの略。
[12]Repli-seq、scRepli-seq
scRepli-seqは、増殖中の細胞でゲノムDNAが倍加していく過程を1細胞レベルで全ゲノム解析する方法。Repli-seqは、scRepli-seq以前に開発された手法で、細胞集団を実験対象としたゲノムDNA複製状態の全ゲノム解析手法。scRepli-seqはsingle-cell DNA Replication sequencingの略。
[13]エピジェネティック修飾
染色体を構成するDNAやヒストンタンパク質への後天的な化学修飾(メチル化、アセチル化など)。塩基配列の変化を伴わずに、遺伝子発現などのゲノム機能を制御する仕組みといわれている。
[14]エスケープ遺伝子
不活性X染色体上にわずかながら存在する転写活性を示す遺伝子。つまり、X染色体不活性化を免れる(エスケープする)遺伝子のこと。
【共同研究グループ】
理化学研究所 生命機能科学研究センター 発生エピジェネティクス研究チーム
チームリーダー 平谷伊智朗(ヒラタニ・イチロウ)
研究員 ラウィン プーンパーム(Rawin Poonperm)
上級研究員 三浦 尚(ミウラ・ヒサシ)
テクニカルスタッフⅠ 谷川明恵(タニガワ・アキエ)
近畿大学 農学部 生物機能科学科 動物分子遺伝学研究室
教授 佐渡敬(サド・タカシ)
大学院生(研究当時) 市原沙也(イチハラ・サヤ)
(現 国立遺伝学研究所 特任研究員)
大阪大学大学院 理学研究科 生物科学専攻 染色体構造機能学研究室
教授 小布施力史(オブセ・チカシ)
准教授 長尾恒治(ナガオ・コウジ)
【研究支援】
本研究は、理化学研究所運営費交付金(生命機能科学研究)で実施し、科学技術振興機構(JST)戦略的創造研究推進事業チーム型研究(CREST)の研究領域「ゲノムスケールのDNA設計・合成による細胞制御技術の創出(研究総括:塩見春彦)」の研究課題「潜在的不安定性から読み解くゲノム設計原理(研究者:平谷伊智朗)JPMJCR20S5」、日本学術振興会(JSPS)科学研究費助成事業若手研究「Inactive-X switching: a novel checkpoint mechanism that sense chromosomal abnormalities?(研究代表者:ラウィン・プーンパーム)」、同挑戦的研究(開拓)「1細胞全ゲノム解析の第二世代化と多次元化への挑戦(研究代表者:平谷伊智朗)」、同新学術領域研究(研究領域提案型)「細胞分化にともなうクロマチンポテンシャルの変化とその分子基盤(研究分担者:平谷伊智朗)」による助成を受けて行われました。
【関連リンク】
農学部 生物機能科学科 教授 佐渡敬(サドタカシ)
https://www.kindai.ac.jp/meikan/1258-sado-takashi.html