虫が残したフンやだ液などに含まれるDNAから植物上にいる多種多様な虫を検出する方法を開発

近畿大学農学部(奈良県奈良市)農業生産科学科講師 米谷 衣代、京都大学白眉センター特定准教授(論文執筆当時、現:香港科技大学教授)潮 雅之、龍谷大学先端理工学部環境生態工学課程教授 三木 健による研究グループは、虫のフンやだ液などに含まれるDNA(環境DNA※1)を用いて、植物上にいる多種多様な虫を検出する方法を開発しました。人工的にかけた水や雨が植物の表面を洗いながら葉や茎を伝って地面に落ちてくるのを集め、そこから環境DNAを取り出すことで、植物上にどのような虫がいたかを明らかにします。本研究成果は、今後、生物多様性の調査や害虫管理への応用が期待されます。
本件に関する論文が、令和5年(2023年)5月12日(金)18:00(日本時間)に、自然科学分野の学術誌"Scientific Reports"にオンライン掲載されました。
【本件のポイント】
●植物上に残された多種多様な虫たちのDNAを植物から一気に収集する方法を開発
●植物表面を流れる水から環境DNAを抽出し、次世代シーケンサーを用いて分析
●生物多様性の調査や農作物等の害虫・天敵の調査への活用に期待
【本件の背景】
昆虫を含む多様な虫たち(節足動物)は、すべての動物の70%以上を占めるといわれ、植物を基盤とする生物の集まり(生物群集)を構成する重要な役割を果たしています。このような虫たちについて調査することは、生物多様性の把握と保全だけでなく、化学農薬を用いない農林害虫防除においても有益な情報となります。しかし、虫たちは微小なものや移動性の高いもの、隠れているものが多いため、それらを一度に調査するにはかなりの労力やコストがかかります。さらに、その多様さゆえに同定が難しく、分類に関する専門的な知識も必要とします。
近年、このような発見しにくい生物や生物多様性の調査において、環境DNAの利用が試みられていますが、水域を対象とする研究が中心で、陸上の植物を利用する生物の研究はあまり進んでいません。その理由の一つとして、水を汲むことで環境DNAを回収できる水域とは異なり、陸上の植物から環境DNAを回収するのは困難であることが挙げられます。先行研究では、植物体の一部(花や食害部位)から環境DNAを回収する方法が発表されていますが、部位や条件が限定されています。
そこで本研究では、特定の部位を対象とせず、また食害跡が見つけられない虫や、どこに隠れているかわからない虫も対象として、植物を傷つけることなく、植物上に残留した環境DNAを収集する方法を開発することを目指しました。
【本件の内容】
研究チームは、陸上の植物を傷つけることなく、環境DNAを収集する方法を開発するにあたり、虫たちが植物上で食事や排泄をする際に排出されるDNAに注目しました。
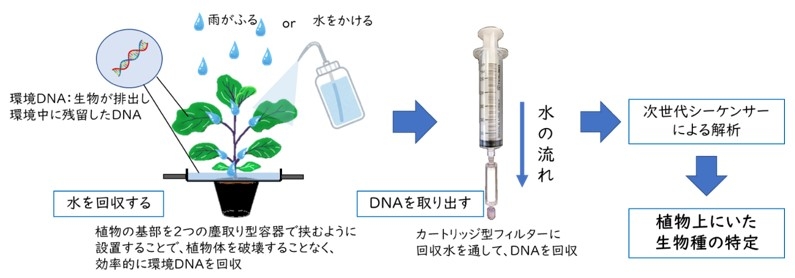
まず、人工的にかけた水や空から降ってくる雨が植物の表面を流れ、葉や茎を伝って地面に落ちてくるのを根もとに設置した容器で収集し、その水から環境DNAを抽出します。次に、抽出した環境DNAについて次世代シーケンサーを用いたメタバーコーディング※2 の手法で分析し、植物上にどのような虫がいたのかを調べます。
この方法によって、人工的にアブラムシを接種した鉢植えのナスと、自然に付いた虫を確認した鉢植えや地植えのキャベツなどを調査したところ、64の節足動物の分類群を科レベルまで同定することができました。このうち種まで特定できたのは29種で、アブラムシやチョウ、ガの幼虫など植物上で視覚的に観察できた、または人工的に導入した7種はすべて含まれていました。また、視覚的調査では確認されなかったチョウやハダニ、バッタ、カマキリなどの22種も検出できました。
なお、研究チームは、この新たに開発した検出方法(特許取得)を動画で公開しています。
https://sites.google.com/view/ecology-method-portal/experiments/eDNA-on-plant
【論文掲載】
掲載誌 :Scientific Reports(インパクトファクター:4.996)
論文名 :Non-destructive collection and metabarcoding of arthropod environmental DNA remained on a terrestrial plant
(陸生植物に残存する節足動物環境DNAの非破壊的収集とメタバーコーディング)
著者 :米谷 衣代1、2、潮 雅之3、4、5、三木 健2、6
所属 :1 近畿大学農学部、2 龍谷大学生物多様性科学研究センター、3 京都大学白眉センター、4 京都大学生態学研究センター、5 香港科技大学海洋学科、6 龍谷大学先端理工学部環境生態工学課程
論文掲載:https://doi.org/10.1038/s41598-023-32862-4
DOI :10.1038/s41598-023-32862-4
【研究の詳細】
研究チームは、人工的にアブラムシを接種した鉢植えのナスと、自然に付いた虫を確認した鉢植えや地植えのキャベツ等から、植物体を傷つけることなく環境DNA(eDNA)を回収しました。eDNAを回収する方法はシンプルで、水道水や滅菌水を植物体に散布したり、雨を利用したりして、植物表面を流れる水を植物の基部に設置した容器で回収しました。研究チームは、この方法を「プラント・フロー・コレクション法(植物流収集法)」と命名しました。その後、収集した水からeDNAを抽出し、シトクロームcオキシダーゼサブユニットI(COI)遺伝子※3 のDNAバーコード領域の塩基配列を、次世代シーケンサーIllumina Miseq※4 のプラットフォームにより解読しました。
その結果、収集した37サンプルにおいて、科まで特定できた分類群は64あり、15目、42科、46属、29種が特定されました。人工的に接種したか、植物に自然に付いていた(目視で観察した)虫は、7種すべてが検出されました。ナスにアブラムシを接種した場合は、植物体に散布するのが水道水でも滅菌水でも、アブラムシのeDNAが100%検出されました。そのほか、自然に付いていた(目視で確認した)虫も含めて、全体的に82%という高い検出率を示しました。また、野外のキャベツから収集したeDNAのサンプルからは、水道水を使った場合は10種、雨を収集した場合は16種と、多くの種が検出できました。さらに、目視で確認した虫以外にも、チョウやアブラムシの寄生バチ、ハダニ、アザミウマ、バッタ、カマキリ等、多種多様な種類の虫を検出しました。これは、目視では見逃した小さな虫や、すでに植物から離れていた虫のeDNAも回収できたことを示しています。
この結果により、本研究で開発したeDNAの収集方法は、水道水、滅菌水、雨を使って、植物を破壊することなく多種多様な虫のeDNAを収集し、検出することが可能であることを示しました。今後、植物上に生息する生物多様性の調査や農作物等の害虫や天敵の調査への活用が期待されます。
【研究者のコメント】
米谷 衣代(よねや きぬよ)
所属 :近畿大学農学部農業生産科学科
近畿大学大学院農学研究科
職位 :講師
学位 :理学(博士)
コメント:本研究は平成28年(2016年)に発案して、平成29年(2017年)から科研費の若手研究(B)の予算をいただいて実行できました。論文の発表までに時間がかかりましたが、今後、多くの方に興味を持っていただき、植物上のeDNAの研究について、研究者だけでなく、一般の方や企業など多くの方々とともに発展させていきたいと考えています。
【研究支援】
本研究は、JSPS科研費 JP 17K15235(米谷 衣代に対して)と、京都大学白眉プロジェクト(潮 雅之に対して)の支援を受けて実施しました。
【用語解説】
※1 環境DNA:生物が水や土や植物などの環境中に排出し、環境中に残留しているDNA。
※2 メタバーコーディング:種同定の指標となる遺伝子領域(DNAバーコード領域)について、次世代シーケンサーを用いて配列決定を行い、科や属、種などの分類群の同定をする方法。大量のサンプルに含まれる複数の種を一度に特定することが可能。
※3 シトクロームcオキシダーゼサブユニットI(COI)遺伝子:この遺伝子はミトコンドリアDNA上に位置しており、昆虫などの真核生物の種を判別するために用いられる。
※4 次世代シーケンサーIllumina Miseq:たくさんのDNA分子を同時に配列決定できるIllumina社の次世代シーケンサー。次世代シーケンシングでは、複数個体を同時に配列決定できる。龍谷大学生物多様性科学研究センター実験室に設置。
【関連リンク】
農学部 農業生産科学科 講師 米谷 衣代(ヨネヤ キヌヨ)
https://www.kindai.ac.jp/meikan/2067-yoneya-kinuyo.html



